Institut de Chimie Physique (ICP)

L’Institut de Chimie Physique (ICP) est une unité mixte de recherche interdisciplinaire ancrée en Chimie, avec des projets aux interfaces avec la Physique et les Sciences du Vivant. L’Unité est structurée autour de quatre groupes scientifiques (RISMAS, CpSysBio, TEMiC, TheoSim) associés à deux plateformes uniques en France :
I) l’accélérateur ELYSE pour la radiolyse et photolyse pulsée ultrarapide et absorption transitoire dans l’UV-Visible;
II) le laser à électrons libres CLIO couplé à la spectrométrie de masse dans l’infrarouge.
L’ICP héberge également des plateaux techniques de pointe de spectro-imagerie résolue en temps basée sur des sources de lumière laser s’étendant sur la gamme spectrale UV-Vis-IR: spectroscopie optique non linéaire visible-infrarouge à deux couleurs (2C-SFG); conductivité microonde résolue en temps dans le visible (TRMC) ; nano-spectro-imagerie dans l’infrarouge (NanoIR) ; spectro-imagerie par fluorescence résolue en temps dans l’UV-visible. L’utilisation d’interaction avec la lumière permet d’étudier des processus chimiques sur de larges gammes temporelles, de la femtoseconde à la seconde.
Faits marquants

Cette couverture de la revue ChemPhysChem illustre le partage dynamique de protons entre les deux extrémités carboxyliques d'un acide dicarboxylique. Les spectres expérimentaux de photodissociation multiple infrarouge (IRMPD) des acides dicarboxyliques déprotonés, HO2 (CH2)n CO2- (n = 2-4), sont interprétés par diverses méthodes de calcul. Les bandes larges près de 1600 cm − 1 ne peuvent être reproduites ni par calculs vibrationnels statiques basés sur la chimie quantique ni par description dynamique de structures individuelles utilisant le champ de force AMOEBA polarisable à plusieurs corps, ce qui suggère que ces molécules subissent un partage dynamique de protons entre les deux extrémités carboxyliques. Pour confirmer cette hypothèse, AMOEBA a été combiné avec un modèle de liaison de valence empirique à deux états (EVB) pour permettre le transfert de protons dans les simulations classiques de dynamique moléculaire. Sur la base d'un paramétrage approprié basé sur des données de référence ab initio, le modèle EVB-AMOEBA reproduit de manière satisfaisante les spectres infrarouges expérimentaux, et la dynamique de température finie révèle une quantité significative de partage de protons dans de tels systèmes.
Référence :
Florian Thaunay, Florent Calvo, Edith Nicol, Gilles Ohanessian, Carine Clavaguéra ; Infrared Spectra of Deprotonated Dicarboxylic Acids: IRMPD Spectroscopy and Empirical Valence‐Bond Modeling. ChemPhysChem 20, 803-814 (2019)
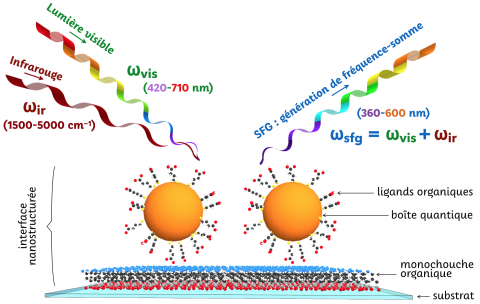
Cette image illustre la démonstration grâce à la spectroscopie par génération de fréquence-somme à deux couleurs (2C-SFG) que des petites molécules greffées (ligands) sur des boîtes quantiques peuvent être détectées dans un champ de lumière intense par amplification de leurs vibrations en tirant parti de l’excitation optique des charges électriques (excitons) confinées au cœur de ces nanocristaux. Ce travail dévoile, contrairement à l’idée dominante, que ce n’est pas seulement l’environnement chimique de ces nanostructures quantiques qui module leurs propriétés optiques fondamentales, mais que l’énergie confinée dans ces boîtes peut être directement transférée à leur entourage sans que celui-ci soit optiquement actif. Ce mécanisme physique fait l’objet d’une étude expérimentale complète qui concorde avec les prédictions du modèle physique développé pour cette dernière, comme publiée dans la revue Communications Chemistry et mis en lumière sur les posts de blog de la Nature Chemistry Community Research et par un communiqué de l’INC du CNRS.
Référence :
T. Noblet, L. Dreesen, S. Boujday, C. Méthivier, B. Busson, A. Tadjeddine et C. Humbert ; Semiconductor quantum dots reveal dipolar coupling from exciton to ligand vibration. Commun Chem 1, 76 (2018).

Cette couverture de la revue ChemBioChem illustre l’optimisation d’une sonde bimodale pour la détection de protéines intracellulaires qui combine la RMN hyperpolarisée et la spectroscopie de fluorescence. Une compréhension complète des phénomènes biologiques implique une détection sensible et non invasive. La partie détection de fluorescence est composée de six résidus contenant un tag tétracystéine (-CCXXCC-) génétiquement incorporé dans la protéine d'intérêt et d'une petite molécule organique, CrAsH. CrAsH devient fluorescent s'il se lie au marqueur tétracystéine. La partie du biocapteur qui permet la détection au moyen de la spectroscopie RMN 129Xe, qui est liée au groupement CrAsH par un espaceur, est basée sur un cœur de cryptophane parfaitement adapté pour héberger du Xénon de manière réversible. Trois peptides différents, contenant le tag tétracystéine et quatre biocapteurs organiques de stéréochimie différente, sont référencés pour proposer le meilleur couple parfaitement adapté à la détection in vitro de protéines.
Référence :
Emilie Mari, Yasmina Bousmah, Céline Boutin, Estelle Léonce, Gaelle Milanole, Thierry Brotin, Patrick Berthault, Marie Erard ; Bimodal Detection of Proteins by 129Xe NMR and Fluorescence Spectroscopy. ChemBioChem 20, 1450-1457 (2019)

